服务介绍
ABclonal 作为科研抗体领域的专业品牌,致力于修饰性抗体的研发。我们经过多年的努力,已经成功研制出了上千种修饰性抗体,并且建立了强大的抗体检测平台,包括 WB、IHC、IF、IP、RIP、ChIP 、ChIP-Seq、shRNA靶点敲除验证等多元化检测手段。我们竭诚为客户提供全面并且可靠的抗体产品。
依托于 ABclonal 抗体库中高质量 ChIP 级别的抗体,结合高通量测序技术,我们推出了ChIP-Seq 特色服务品牌 Omicgene。我们主要技术团队在表观遗传学领域有丰富的研发经验,自品牌建立以来已立项近千个。目前,我们是国内少有的能够提供 ChIP 实验、ChIP-qPCR 检测、高通量测序文库构建、生物信息学数据分析全套服务的技术外包服务团队。

ChIP-Seq 原理
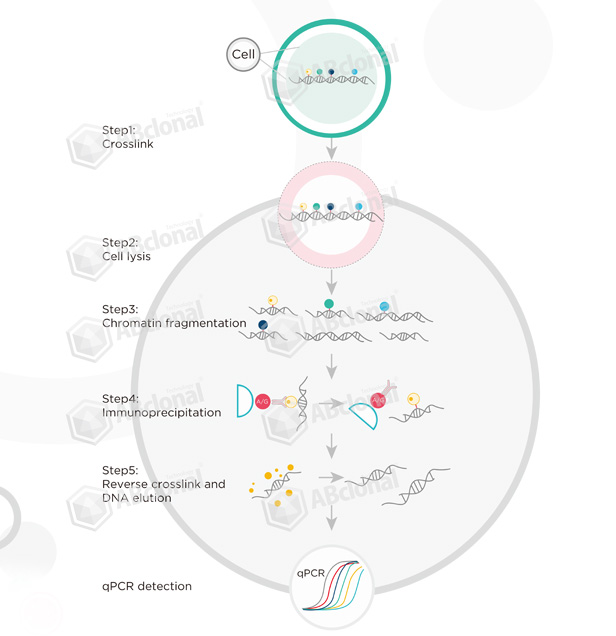
- 染色质免疫共沉淀(Chromatin Immunoprecipitation,ChIP)技术主要是用于研究 DNA 和蛋白质的相互作用。染色质主要是由组蛋白和缠绕在上面的 DNA 组成。在染色质上还结合了许多转录因子、染色质重塑相关蛋白和非编码 RNA。染色质上结合的蛋白或者组蛋白上不同的共价修饰可以通过改变染色质的结构,或者招募相关的因子来调控基因的表达。通过 ChIP 技术,我们可以研究不同的组蛋白修饰、转录因子以及染色质上结合的一些其他蛋白,在基因组上的分布。
- ChIP 实验的主要原理是,当 DNA 和蛋白结合或者距离很近时,通过甲醛固定交联,在 DNA 和蛋白分子之间形成共价键。将染色质通过超声波破碎或者微球菌核酸酶处理,打断成小片段,然后通过特异性的 ChIP 级别抗体,富集目的蛋白。经过逆交联处理之后,消化水解目的蛋白,回收得到目的 DNA,从而得到与目的蛋白相互作用的 DNA 信息。
- ChIP-Seq 技术是将传统的 ChIP 实验与高通量测序技术结合,对 ChIP 实验富集得到的目的 DNA 进行高通量测序分析,得到全基因范围目的蛋白结合的序列信息和位置信息。
技术优势
- 全面的 ChIP 级别抗体库
- 经验丰富的实验团队
- 专业的数据分析团队
- 多组学联合、个性化分析
- 依托于武汉大学的技术团队